Home
Программа работает в среде MS DOS (используйте DosBox с русификатором russian.txt).
Если работает очень медленно или зависает, откройте параметры (F4) и измените Delay на 1. Также, можете дополнительно изменять количество циклов DosBox (Ctrl+F11 / Ctrl+F12).
Данная программа была создана для демонстрации и изучения закономерностей
протекания катализируемых ферментами реакций, осуществляемой при
помощи набора систем уравнений, являющихся ядром программы. Каждая
кинетическая модель передает программе номер нужной ей cистемы
уравнений и параметры к ним. Таким образом, на основании одной
системы уравнений может быть создано большое количество разнообразных
кинетических моделей, демонстрирующих основные особенности данного
типа взаимодействий.
Cодержание справки:
1. Управление программой
2. Структура файлов экосистем
3. Уравнения и модели
4. Замечание об ошибках
5. Источники
-
Управление программой:
В режиме выбора и редактирования экосистемы:
F1 - Вызов этой справки
F4 - Редактирование параметров выбранной экосистемы или смена
описания папки
F5 - копирование выбранной экосистемы
F6 - перемещение выбранной экосистемы
F7 - создание новой папки
Ins - создание новой экосистемы
Del - удаление папки/экосистемы
BkSp - возврат в предыдущую папку
Enter - переход в режим моделирования и создание графика числен-
ностей и концентраций для выбранной экосистемыВ режиме моделирования экосистемы:
Esc - Возврат в режим выбора и редактирования
Enter - Продолжение моделирования с начала экрана
P - Приостановка моделирования
Tab - Включение / выключение вывода параметров моделируемой
системы.
F1 - Включение / выключение вывода комментариев по данной
модели.
При нажатии любой клавиши кроме Enter и P моделирование начина-
ется с начала (с начальных параметров) -
Программа содержит несколько уравнений моделирования популяций. В файлах
систем номер системы является самым первым параметром (Type).
В поставляемых экосистемах после заголовка следуют уравнения дан-
ного Type'a. Если вы создаете свой файл, то нужно следовать следу-
ющим правилам:
1. Последовательность параметров должна быть всегда определен-
ной: Type, отметка о выводимых численностях и концентрациях
(Show=xxox), состоящая из x-выводить и o-не выводить,
время (T), шаг интергирования (dT; т.е. для интегрирования
данной системы будет осуществлено T/dT шагов), переключатель
общего/частного максимума по Y (ОбщаяY),
максимальная отображаемая численность (MaxY),
начальные количества каждой популяции (No1,
No2,..), специальные параметры экосистемы. К специальным па-
раметрам (также в определенной последовательности) относят-
ся коэффициенты, подставляемые в уравнения динамик популя-
ций. Переключатель общего/частного максимума сообщает прог-
рамме, следует ли ей читать из файла MaxY для каждого N
перед каждым No (кроме No1 - в этом случае MaxY1=MaxY) или
MaxY является общим масштабным коэффециентом для всех N.
(Примеры: см. поставляемые экостистемы).
P.S. Совершенно не обязательно придерживаться используемых в
поставляемых системах названий (Type=2). Вы можете ис-
пользовать любые названия (в том числе, русские), содер-
жащие любые символы кроме "=".
2. Название перед каждым параметром необязательно, но перед каж-
дым должен стоять знак равенства, иначе строка рассматрива-
ется как комментарий.
3. Все строки файла, не содержащие знака равенства, считаются
комментариями. Но если вы хотите использовать знак равенства
в своем комментарии, начните его с "> ". Кроме того, только
строки, начатые с этой аббревиатуры, выводятся на экран при
нажатии клавиши F1 в режиме моделирования. Комментарии можно
располагать в любом месте файла системы после заголовка.
4. В системе предусмотрено менять названия осей. Изначально оси
X присвоено название "t", оси Y - "N". Смена осуществляется
следующей командой в файле: "# Y=C" или "# X=pH". Эти команды
можно включать в любое место файла после заголовка. -
Каждому Type соответствует определенный набор уравнений динамики концен-
траций веществ системы и параметров:
1. Рассчет начальной стационарной скорости на основе уравнения
Михаэлиса в зависимости от начальных концентраций субстрата.
- kk - каталитическая константа (равна максимальной скорости
реакции, деленной на начальную концентрацию фермента:
kk=Vm/Eo )
- Eo - начальная концентрация фермента
- Km - константа Михаэлиса (в схеме Михаэлиса равна отношению
суммы констант разрушения фермент-субстратного комп-
лекса с образованием субстрата/продукта к константе
образования фермент-субстратного комплекса (ФСК):
Km=(k1+k2)/k3 )
- k - коэффициент роста субстрата (коэф. пропорциональности)
Вывод: Vo, So
Реакции: Km kk
E+S <-> ES -> E+P
Уравнения: Vo=(kkEoSo)/(Km+So) (скорость)
dSo=k (субстрат)
Параметры: kk, Eo, Km, k
Пример: STAT\VS\vs
2. Рассчет начальной стационарной скорости на основе уравнения
Михаэлиса в зависимости от начальных концентраций субстрата
в условиях ингибирования (активации) избытком субстрата.
На основе модели N 1.
- a - коэффициент активации (a>1) или ингибирования (a<1).
a=0 означает, что "тройной комплекс" фермента с двумя
молекулами субстрата не реакционноспособен
Вывод: Vo, So
Реакции: Km kk
E+S <-> ES -> E+P
Ki akk
ES+S <-> ES2 -> ES+P
Уравнения: Vo=((1+aSo/Ki)kkEoSo)/(Km+So+SoSo/Ki)
(скорость)
dSo=k (субстрат)
Параметры: kk, Eo, Km, k, a, Ki
Пример: STAT\VS\vsiis
3. Рассчет насыщения переносчика субстратом (4 активных центра).
Например: насыщение гемоглобина кислородом. Данную стацио-
нарную модель можно также использовать для рассчета отношения
концентрации фермент-субстратного комплекса к концентрации
самого комплекса. На основе модели N 1.
- Kr, Kt - эффективные константы связывания, являющиеся функ-
циями констант равновесия в схеме
- L - константа равновесия между P и P'
Вывод: a, S
Реакции: L
P <-> P'
P+S <-> PS P'+S <-> P'S
PS+S <-> PS2 P'S+S <-> P'S2
PS2+S <-> PS3 P'S2+S <-> P'S3
PS3+S <-> PS4 P'S3+S <-> P'S4
Уравнения: a=(S/Kr(1+S/Kr)^3+LS/Kt(1+S/Kt)^3)/
(L(1+S/Kt)^4+(1+S/Kr)^4) (скорость)
dSo=k (субстрат)
Параметры: kk, Eo, Km, k, a, Ki
Пример: STAT\HBO\hbo
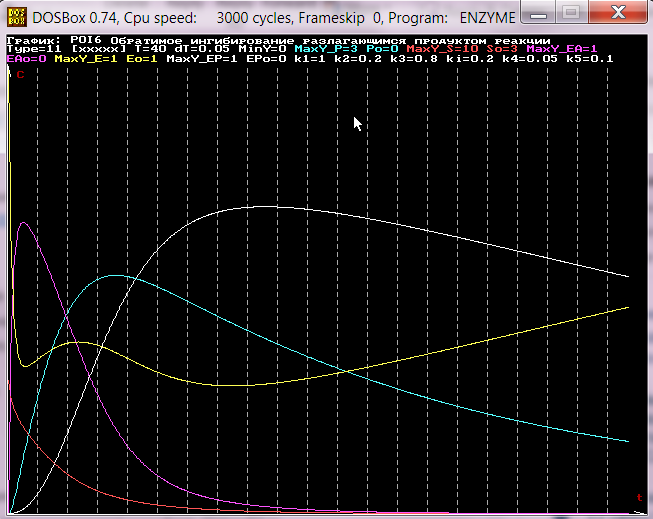
4. Модель ферментативных реакций, один из продуктов которых
образуется на стадии формирования фермент-субстратного
комплекса. Например, сериновые протеазы образуют ацилфермент
и спирт (амин). На основе модели N 5.
Вывод: P1, E, S, EA, P2
Реакции: k1 k2
E+S -> P1+EA -> E+P2
Уравнения: dP1=k1ES (первый продукт)
dE=k2EA-k1ES (фермент)
dS=-k1ES (субстрат)
dEA=-k2EA+k1ES (ацилфермент)
dP2=k2EA (второй продукт)
Параметры: k1, k2
Пример: NSTAT\AF*
NSTAT\AF\IS*
5. Модель типичной ферментативной реакции по схеме Михаэлиса.
- k(n) - константы превращений
Вывод: P, S, ES, E
Реакции: k1,k2 k3
E+S <-> ES -> E+P
Уравнения: dP1=k3ES (продукт)
dS=k2ES-k1ES (субстрат)
dES=k1ES-k2ES-k3ES (ФСК)
dE=k2ES-k1ES (фермент)
Параметры: k1, k2, k3
Пример: NSTAT\SIMPLE*
NSTAT\SIMPLE\IS*
6. Рассчет зависимости скорости реакции от pH.
- Ka, Kb - константы кислотности и основности фермента
- Ka', Kb' - константы кислотности и основности ФСК
Вывод: kk, Km, [H], v
Уравнения: kk=k2/(1+[H]/Ka'+Kb'/[H])
(каталитическая константа)
Km=Ks(1+[H]/Ka+Kb/[H]) (константа Михаэлиса)
dpH=dT (кислотность среды)
v=kkEoSo/(Km+So) (скорость)
Параметры: k1, k2, Ka, Ka', Kb, Kb', Eo, So
Пример: STAT\PH*1. Элементарная модель лимитирования по субстрату без учета ги- бели клеток. - Ys - экономический коэффициент (определяет, сколь- ко субстрата поглощает одна клетка за единицу времени) - Mm - предельная максимальная удельная скорость роста. - Ks - параметр, характеризующий сродство субстрата к клет- кам культуры Популяции: 1 (3) Уравнения: dS=-N/Ys (субстрат) m=Mm*S/(Ks+S) (скорость роста) dN=N*m (число клеток) Параметры: Ys, Mm, Ks Пример: 1\SUBSTRAT\simple* 2. Логистическая модель лимитирования по субстрату с учетом гибели клеток. На основе модели N 1. - M1 - "ресурсный" коэффициент. Ограничивает максимальное число клеток: Nm=M1/Mm Популяции: 1 (3) Уравнения: dS=-N/Ys (субстрат) m=Mm*S/(Ks+S) (скорость роста) dN=N*m-N*N*M1 (число клеток) Параметры: Ys, Mm, Ks, M1 Пример: 1\SUBSTRAT\logist* 3. Логистическая модель лимитирования по субстрату с учетом ингибирования избытком субстрата. При начальных больших количествах субстрата штамм вымирает, при средних коли- чествах рост культуры в начале немного ингибируется, но, постепенно снижая концентрацию субстрата, бактерии постепенно уменьшают силу ингибирования. На основе модели N 2 - Ki - константа ингибирования избытком субстрата Популяции: 1 (3) Уравнения: dS=-N/Ys (субстрат) m=Mm*S/(Ks+S*(1+S/Ki)) (скорость роста) dN=N*m-N*N*M1 (число клеток) Параметры: Ys, Mm, Ks, Ki, M1 Пример: 1\SUBSTRAT\sii* 4. Логистическая модель ингибирования субстратом при линейном контролируемом повышении его концентрации. Фактически эта "модель" выводит на экран зависимость скорости роста клеток от концентрации субстрата в среде. На основе модели N 3 - Sk - скорость роста субстрата Популяции: 1 (3) Уравнения: dS=Sk (субстрат) m=Mm*S/(Ks+S*(1+S/Ki)) (скорость роста) dN=N*m-N*N*M1 (число клеток) Параметры: Sk, Mm, Ks, Ki, M1 Пример: 1\SUBSTRAT\siim* 5. Логистическая модель ингибирования стабильным продуктом жиз- недеятельности клеток. На основе модели N 2 - Ki - константа ингибирования продуктом - Yp - количество продукта на единицу биомассы Популяции: 1 (4) Уравнения: dS=-N/Ys (субстрат) m=Mm*S/(Ks*(1+P/Ki)+S) (скорость роста) dN=N*m-N*N*M1 (число клеток) dP=N/Yp (продукт) Параметры: Ys, Mm, Ks, Ki, M1, Yp Пример: 1\INGIB\pi* 6. Логистическая модель ингибирования роста клеток продуктом их жизнедеятельности, разрушающимся по реакции 1 порядка. На основе модели N 5 - Dp - константа скорости реакции разрушения продукта Популяции: 1 (4) Уравнения: dS=-N/Ys (субстрат) m=Mm*S/(Ks*(1+P/Ki)+S) (скорость роста) dN=N*m-N*N*M1 (число клеток) dP=N/Yp-Dp*P (продукт) Параметры: Ys, Mm, Ks, Ki, M1, Yp, Dp Пример: 1\INGIB\pid* 7. Логистическая модель лимитирования роста клеток по линейно возобновляющемуся субстрату. Субстрат буквально добавляется в систему с постоянной скоростью. На основе модели N 2 - Sd - скорость добавления субстрата Популяции: 1 (3) Уравнения: dS=-N/Ys+Sd (субстрат) m=Mm*S/(Ks+S) (скорость роста) dN=N*m-N*N*M1 (число клеток) Параметры: Ys, Mm, Ks, M1, Sd Пример: 1\SUBSTRAT\sr* 8. Модель одного из механизмов индукции. Активное размножение клеток не начинается до тех пор, пока концентрация ингиби- тора роста не станет достаточно низкой. (логистическая) На основе модели N 6 Популяции: 1 (4) Уравнения: dS=-N/Ys (субстрат) m=Mm*S/(Ks*(1+З/Ki)+S) (скорость роста) dN=N*m-N*N*M1 (число клеток) dP=Dp*P (ингибитор) Параметры: Ys, Mm, Ks, Ki, M1, Dp Пример: 1\INGIB\psd* 9. Логистическая модель развития и гибели штамма в результате исчерпания субстрата и постепенной инактивации способных к делению клеток. На основе модели N 2 - L - константа скорости инактивации клеток Популяции: 1 (4) Уравнения: dS=-N/Ys (субстрат) m=Mm*S/(Ks*(1+S/Ki)+S) (скорость роста) dI=A*L-I*I*M1 (неактивные клетки) dA=A*m-A*A*M1 (активные клетки) N=I+A (число клеток) Параметры: Ys, Mm, Ks, L, M1 Пример: 1\STOP\ia* 10. Логистическая модель развития и гибели штамма в результате инактивации клеток по "запрограммированному отказу". В мо- дели предполагается, что скорость размножения клеток прямо пропорциональна числу рецепторов к фактору роста на клетках: R=A*[Exp(-Lt)-Exp(-at)]. На основе модели N 2 - L - константа скорости потери рецепторов, инактивации - a - константа скорости роста количества рецепторов - A - коэффициент пропорциональности Популяции: 1 (4) Уравнения: dz1=-z1*L (дополнительные dz2=-z2*a функции) m=A*(z1-z2) (скорость роста) dN=N*m-N*N*M1 (активные клетки) Параметры: L, a, A, M1 Пример: 1\STOP\zo* 11. Модель культивирования клеток в режиме хемостата. Неослож- ненный рост. Коэффициент D показывает скорость вымывания ве- щества и культуры из ферментера, то есть скорость разбавле- ния раствора с культурой раствором с субстратом. На основе модели N 2 - D - скорость разбавления - Se - концентрация субстрата во входящем растворе Популяции: 1 (4) Уравнения: m=Mm*S/(Ks+S) (скорость роста) dN=N*m-N*D (число клеток) dS=D*(S-Se)-m*N/Ys (субстрат) dP=m*N/Yp-P*D (продукт) Параметры: Ys, Mm, Ks, D, Se, Yp Пример: 1\STAT\simple* 12. Модель культивирования клеток в режиме хемостата. Рост клеток ингибируется продуктом их жизнедеятельности. На основе модели N 11, 5. Необходимо помнить, что все компонен- ты системы постепенно вымываются из нее, в том числе и продукт жизнедеятельности, что и обеспечивает возможность размножения. Популяции: 1 (4) Уравнения: m=Mm*S/(Ks*(1+P/Ki)+S) (скорость роста) dN=N*m-N*D (число клеток) dS=D*(S-Se)-m*N/Ys (субстрат) dP=m*N/Yp-P*D (продукт) Параметры: Ys, Mm, Ks, D, So, Yp, Ki Пример: 1\STAT\pic* 13. Модель лимитирования по субстрату с учетом лизиса клеток 1 порядка. На основе модели N 1 - M1 - константа скорости лизиса Популяции: 1 (3) Уравнения: dS=-N/Ys (субстрат) m=Mm*S/(Ks+S) (скорость роста) dN=N*m-N*M1 (число клеток) Параметры: Ys, Mm, Ks, M1 Пример: 1\SUBSTRAT\lisis* 14. Логистическая модель динамики численностей двух клеточных популяцей, концентраций их субстратов и продуктов. Популя- ции взаимодействуют по принципу симбиотрофной ассоциации, т.е. продукт жизнедеятельности клеток одного вида является субстратом для клеток другого вида. На основе модели N 2. Популяции: 2 (7) Уравнения: dS1=-N1/Ys1 (субстрат-1) m1=Mm1*S1/(Ks1+S1) (скорость-1) dN1=N1*m1-N1*N1*M1 (клетки-1) dS2=-N2/Ys2+N1/Yp1 (продукт-1/ субстрат-2) m2=Mm2*S2/(Ks2+S2) (скорость-2) dN6=m2*N2-N2*N2*M2 (клетки-2) dP2=N2/Yp2 (продукт-2) Параметры: Ys1, Mm1, Ks1, M1, Yp1, Ys2, Mm2, Ks2, M2, Yp2 Пример: 2\* 15. Логистическая модель динамики численностей трех клеточных популяцей, концентраций их субстратов и продуктов. Популя- ции взаимодействуют по принципу симбиотрофной ассоциации, т.е. продукт жизнедеятельности клеток одного вида является субстратом для клеток другого вида, а продукт жизнедеятель- ности последнего является субстратом для третьего вида. На основе модели N 14. Популяции: 3 (10) Уравнения: dS1=-N1/Ys1 (субстрат-1) m1=Mm1*S1/(Ks1+S1) (скорость-1) dN1=N1*m1-N1*N1*M1 (клетки-1) dS2=-N2/Ys2+N1/Yp1 (продукт-1/ субстрат-2) m2=Mm2*S2/(Ks2+S2) (скорость-2) dN6=m2*N2-N2*N2*M2 (клетки-2) S3=-N3/Ys3+N2/Yp2 (продукт-2/ субстрат-3) m3=Mm3*S3/(Ks3+S3) (скорость-3) dN3=m3*N3-N3*N3*M3 (клетки-3) dP3=N3/Yp3 (продукт-3) Параметры: Ys1, Mm1, Ks1, M1, Yp1, Ys2, Mm2, Ks2, M2, Yp2, Ys3, Mm3, Ks3, M3, Yp3 Пример: 3\* -
В программе использована специальная многоуровневая система контроля
ошибок. Поэтому практически ничто не может самопроизвольно завер-
шить программу без желания пользователя. Если в модель были введе-
ны неестественные коэффициенты, то результат может выйти за пределы
(0.5e4932). В этом случае программа выдаст звуковой сигнал и оста-
новит моделирование, поскольку никаких разумных результатов все равно
уже нельзя будет получить, исходя из данных параметров. После нажа-
тия клавиши программа вернется в меню и выдаст сообщение об ошибке,
предлагая пользователю продолжить работу. Отказываться от продолже-
ния работы имеет смысл только в том случае, если программа цикличес-
ки выдает данную ошибку уже несколько раз и не продолжает работу. Та-
кое в принципе возможно только в случае грубых физических повреждений
компьютера или изменений программы в результате занесения вируса или
попыток изменения кода. Если программа обнаружит заражение вирусом,
она попытается излечить себя с разрешения пользователя, если это ока-
жется возможным. -
При составлении программы и подготовке систем были использованы следующие
источники:
1. С.Д.Варфоломеев, К.Г.Гуревич. "Биокинетика". Москва, "Пранд",
1999
2. Ю.А.Ершов, В.А.Попков, А.С.Берлянд, А.З.Книжник,
Н.И.Михайличенко, "Биофизическая химия". Москва, "Высшая
школа", 1993
3. А.Н.Герасимов, "Математические модели в биологии, экологии и
медицине. Москва, МИФИ, 1998
4. А.О.Рувинский, Л.В.Высоцкая, С.М.Глаголев и др., "Общая биоло-
гия". Москва, "Просвещение", 1993
Со всеми вопросами и предложениями просьба обращаться:
e-mail: rualark@gmail.com
Архипенко Алексей
Москва
1999
Project Members:
- Alexey Arkhipenko (admin)
